La incorporación de la metodología PCR multiplex como apoyo al laboratorio clínico para la detección de patógenos en muestras clínicas obtenidas a partir de diversos cuadros infecciosos, representa un hito que ha contribuido significativamente a lograr un diagnóstico acertado en menor tiempo. Así mismo, se ha demostrado que integrar la detección de genes de resistencia a los antibióticos usualmente utilizados para estos síndromes, mejora la eficiencia y el enfoque de los tratamientos de acuerdo con el perfil completo del microorganismo y no solo a su expresión fenotípica.
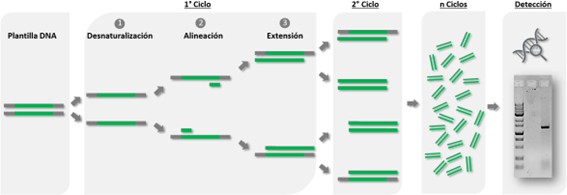
La metodología PCR multiplex (reacción en cadena de la polimerasa) permite detectar el material genético de diversos microorganismos que pueden estar presentes en muestras clínicas específicas obtenidas a partir de una infección localizada. Gracias a sus múltiples bondades, como una mayor sensibilidad analítica respecto a metodologías convencionales que dependen del metabolismo bacteriano, detección de coinfecciones, proceso a partir de una única muestra y el uso de una sola prueba, se logra disminuir el tiempo de respuesta del servicio, las intervenciones requeridas por parte del profesional del área, las pruebas adicionales para identificar patógenos que no son cultivables, costos institucionales, tasas de mortalidad y contribuye al control de infecciones intrahospitalarias.
Como resultado de esta incorporación en apoyo diagnóstico, el nuevo reto se encuentra ahora enfocado en:
- El diseño de los ensayos y la capacidad de las plataformas para detectar genes de resistencia a antibióticos adquiridos a partir de fallas terapéuticas y del uso inadecuado de los mismos.
- La calidad y utilidad de las bibliotecas de genes que se encuentren disponibles.
- Fomentar la correlación con la expresión fenotípica obtenida por metodologías convencionales vs. genes de resistencia reportados por PCR.
En los últimos años, la resistencia a los antibióticos ha aumentado de manera gradual generando anualmente a nivel mundial hasta 5 millones de muertes, 3.4 billones de dólares de pérdidas económicas con escala hasta el 2030 y representando una de las 10 principales amenazas a la salud global. La lucha contra la resistencia a los antibióticos reviste una alta prioridad debido a que estas a menudo generan en el ámbito hospitalario una sobrecarga de costos en la atención sanitaria e ineficiencia en las terapias antibióticas implementadas.

Por ejemplo, en Colombia desde hace un tiempo se ha presentado un aumento drástico en la detección de diferentes genes de resistencias. Según el informe Nacional de la Vigilancia de la resistencia Bacteriana del 2022, en las unidades de cuidando intensivo UCI se ha evidenciado un aumento importante en la identificación de enterobacterales productores de Betalactamasas tanto en unidades de adultos como en pediátricas y estos datos han encendido las alarmas dado a la alta mortalidad que representan para estos pacientes cuando se adquieren infecciones por microorganismos multirresistentes. Además de esto, reportan datos preocupantes, debido al aumento de resistencia a los carbapenémicos, siendo estas uno de los mecanismos de resistencias más importantes y de mayor vigilancia debido a que disminuye las opciones terapéuticas e incrementan las estancias hospitalarias, la mortalidad y los costos de la atención.
En pro de esta situación que se ha presentado a nivel global, la OMS propuso un plan de acción para hacerle frente y reducir la tasa de resistencia a los antibióticos. Dentro de estas se encuentran: reforzar la vigilancia activa y la investigación para la detección de genes de resistencia, optimizar el uso de los medicamentos antimicrobianos y mejorar la sensibilización y los conocimientos en materia de resistencias, actividades que involucran de manera activa a los laboratorios clínicos, médicos, PROA, comité de infecciones y entes de vigilancia epidemiológica.
A partir de este plan y la necesidad de contar con tecnologías que contribuyan a la detección de genes de resistencia de microorganismos presentes en diferentes síndromes, se han diseñado pruebas basadas en PCR que permiten realizar esta tarea en menor tiempo y con todas las bondades que representan esta metodología; brindando información adicional que impacta de forma directa en la decisión médica para el tratamiento de los pacientes.
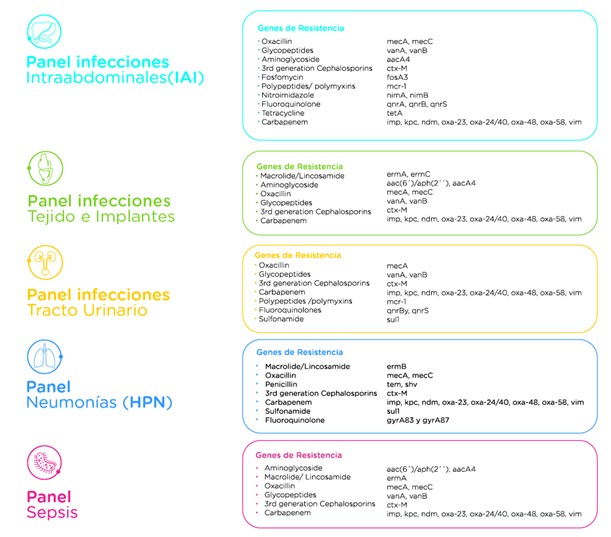
Dentro de las tecnologías desarrolladas, se encuentra el dispositivo médico Unyvero que cuenta con 5 paneles (siendo 3 de innovación) que apuntan a detectar la etiología infecciosa de diferentes síndromes como:
- Infecciones de tejido óseo e implantes
- Infecciones intraabdominales
- Infecciones de tracto urinario
- Neumonía intrahospitalaria
- Sepsis
Dichos paneles, cuentan con una biblioteca amplia de sondas específicas para la detección de microorganismos clínicamente relevantes, genes de resistencia a los antibióticos usualmente empleados para su tratamiento y toxinas; además, permiten utilizar diferentes muestras clínicas que hayan sido obtenidas a partir de estas infecciones, abriendo las posibilidades de lograr un diagnóstico acertado.
Finalmente y por las razones expuestas, Unyvero ha marcado la diferencia gracias al apoyo relevante que ha generado en la detección rápida de microorganismos y genes de resistencia asociados, promoviendo una vigilancia epidemiológica activa y una respuesta rápida en el control de la generación y propagación de resistencias a los antimicrobianos.
¿Quieres conocer más sobre la tecnología Unyvero?
Consulta con tu ejecutivo de ventas o escríbenos a serviciocliente@annardx.com
Bibliografía
- Resistencia a los antibióticos, OMS, 2020. Link de consulta: https://www.who.int/es/news-room/fact-sheets/detail/antibiotic-resistance#:~:text=Estas%20bacterias%20farmacorresistentes%20pueden%20causar,y%20que%20aumente%20la%20mortalidad
- Protocolo de Vigilancia de Resistencia bacteriana a los antimicrobianos en al ámbito hospitalario, INS, 2022. Link de consulta: https://www.ins.gov.co/buscador-eventos/Lineamientos/Pro_Resistencia%20bacteriana%202022.pdf
- Plan Nacional de Respuesta a la Resistencia a los Antimicrobianos, Dirección de Medicamentos y Tecnologías en Salud, Ministerio de Salud y Protección Social, 2018. Link de consulta: https://www.minsalud.gov.co/sites/rid/Lists/BibliotecaDigital/RIDE/VS/MET/plan-respuesta-resistencia-antimicrobianos.pdf
- Lineamientos distritales para la vigilancia y contención de los microorganismos productores de carbapenemasas en las instituciones de salud, ACIN, Secretaria Distrital de Salud de Bogotá, 2022. Link de consulta: https://www.saludcapital.gov.co/DSP/Resistencia%20Bacteriana/VIGILANCIA%20MPC/Lineamientos_distrital_Vig_Carb.pdf
- © Curetis GmbH, 2024 | Manual de la aplicación Unyvero HPN | º art. 00256 Rev. 7.0 – ES
- © Curetis GmbH, 2022 | Manual de la aplicación Unyvero BCU | N.o art. 00344 Rev. 5.0 – ES – G1
- © Curetis GmbH, 2022 | Manual de la aplicación Unyvero ITI | No art. 00230 Rev. 8.0
- © Curetis GmbH, 2022 | Manual de la aplicación Unyvero IAI | N.º art. 00363 Rev. 4.0 – ES – G1
- © Curetis GmbH, 2022 | Manual de la Aplicación Unyvero UTi | n.º art 00537 Rev. 3.0 – ES – G1